最近,加拿大多伦多科学家以前所未有的细节,记录了细胞内所有蛋白质的位置。这种新的蛋白图,可让科学家们更深入地了解“当疾病来袭时细胞内发生了什么”,也将有助于我们找到更好的治疗方法。这项研究发表在六月四日出版的权威杂志《细胞》(Cell)。
在多伦多大学Donnelly中心Brenda Andrews、Charles Boone和Jason Moffat教授的带领下,该研究小组创建了一种最先进的自动化流程,来监测蛋白质位于细胞内什么位置,并探讨它们如何移动来响应遗传或环境变化。
也是在这个月,研究人员可通过美国遗传学会官方杂志《G3: Genes|Genomes|Genetics》,获取这个详细的蛋白质位置数据库,所以任何人都可以查看他们感兴趣的蛋白质的位置和运动。
当细胞在发挥它们作用的时候——如形成、维护和修复我们的身体,它们不断地四处移动蛋白质。但是科学家们对于我们细胞内这一交通是如何发生的,却知之甚少。船运或航空路线,可让我们深入了解世界经济的状态,同样地,这种新的蛋白质图,将帮助科学家更好地了解“当细胞健康时发生了什么,以及生病时发生了什么故障”。
蛋白质是基因的产物,它们负责细胞内的所有工作。Brenda Andrews教授指出,很多调控发生在细胞内,这对于人体的基本功能至关重要,影响着个体蛋白质定位在哪里,以及它们如何移动。如果我们能够理解“为什么细胞是健康的,为什么它们有时会发生病变”,则对于了解这种调控如何发生,将是非常重要的。
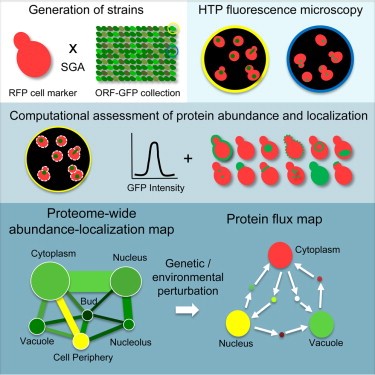
为了可视化和计算细胞中大多数的蛋白质(大约6000个),研究人员了收集了数量惊人的2000多万个细胞的数据。十多年来,科学家们与机械工程师(他们构建机器来处理细胞)和软件编写人员(设计基于人工智能的算法)密切合作,来处理大量数据。
Andrews使用面包酵母作为模型,来理解人类细胞生物学。他说,我们需要在大规模上这样做的原因,是因为实在是有太多的蛋白质。酵母细胞发挥作用的方式非常类似于人类细胞,但有较少的蛋白质,数量大约是更复杂人类细胞中蛋白质的大约四分之一。这种相对简单性,可让Andrews和Boone这样的研究人员利用酵母,对“酵母和人类细胞如何起作用”获得许多基本的见解。
他们的团队不仅记录了正常细胞内蛋白质的运动和丰度,而且也着眼于当细胞携带突变(这可能会导致遗传性疾病),或者接触到不同药物的时候,发生了什么。Andrews说,我们开发的这种方法,可让科学家们检测细胞内所有的蛋白质,以及它们如何改变以响应任何变化。
接下来,研究人员将使用这种强大的流程,来研究蛋白质在人体细胞中如何移动,如癌细胞,以更好地了解疾病的成因,同时也寻求新的治疗方法。
Moffat表示,我们想在系统水平上了解所有蛋白质都是如何移动的,例如,在用药物治疗的癌细胞中,使我们能够根据蛋白质的定位和丰度,识别癌细胞中的漏洞,并开始考虑如何最好地靶定这些变化。
原文标题:Yeast Proteome Dynamics from Single Cell Imaging and Automated Analysis
原文摘要:Proteomics has proved invaluable in generating large-scale quantitative data; however, the development of systems approaches for examining the proteome in vivo has lagged behind. To evaluate protein abundance and localization on a proteome scale, we exploited the yeast GFP-fusion collection in a pipeline combining automated genetics, high-throughput microscopy, and computational feature analysis. We developed an ensemble of binary classifiers to generate localization data from single-cell measurements and constructed maps of ∼3,000 proteins connected to 16 localization classes. To survey proteome dynamics in response to different chemical and genetic stimuli, we measure proteome-wide abundance and localization and identified changes over time. We analyzed >20 million cells to identify dynamic proteins that redistribute among multiple localizations in hydroxyurea, rapamycin, and in an rpd3Δ background. Because our localization and abundance data are quantitative, they provide the opportunity for many types of comparative studies, single cell analyses, modeling, and prediction.